BACK END/R
[R] R 정리 18 - svm, knn
circle kim
2021. 2. 2. 13:16
# SVM 분류 모델 : 데이터 분류 결정 마진에 영향을 주는 관측치(Support Vectors)를 이용
# Kernel trick을 이용해 비선형 데이터에 대한 분류도 가능
library(e1071)
# train/test 생략
head(iris, 3)
# Sepal.Length Sepal.Width Petal.Length Petal.Width Species
# 1 5.1 3.5 1.4 0.2 setosa
# 2 4.9 3.0 1.4 0.2 setosa
# 3 4.7 3.2 1.3 0.2 setosa
x <- subset(iris, select = -Species)
y <- iris$Species
x
ysvm_model <- svm(Species ~ ., data=iris)
svm_model
# Parameters:
# SVM-Type: C-classification
# SVM-Kernel: radial
# cost: 1
#
# Number of Support Vectors: 51
summary(svm_model)
svm_model1 <- svm(x, y)
summary(svm_model1)
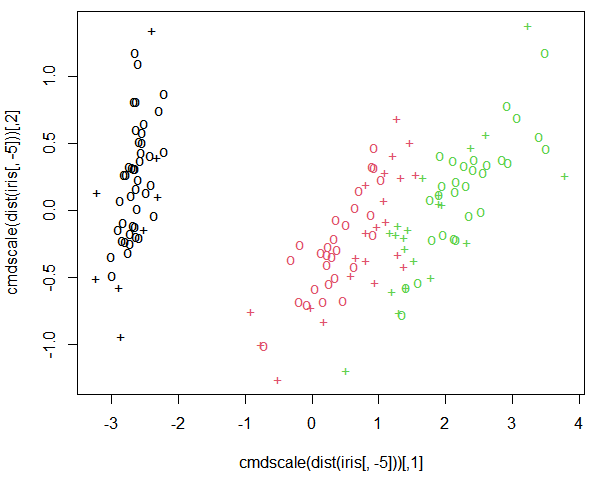
dist(iris[, -5])
cmdscale(dist(iris[, -5])) # 거리 값을 공간적인 배치형식으로 출력
# [,1] [,2]
# [1,] -2.684125626 0.319397247
# [2,] -2.714141687 -0.177001225
# [3,] -2.888990569 -0.144949426
# [4,] -2.745342856 -0.318298979
# [5,] -2.728716537 0.326754513
# [6,] -2.280859633 0.741330449
# [7,] -2.820537751 -0.089461385
# [8,] -2.626144973 0.163384960
# support vector : + , 나머지 데이터는 o
plot(cmdscale(dist(iris[, -5])), col=as.integer(iris[,5]), pch=c("o","+")[1:150 %in% svm_model$index + 1])
pred <- predict(svm_model, x)
head(pred)
t <- table(pred, y)
t
# y
# pred setosa versicolor virginica
# setosa 50 0 0
# versicolor 0 48 2
# virginica 0 2 48
sum(diag(t)) / nrow(x) # 분류 정확도 : 0.9733333# 최상의 모델을 만들기 위해 파라미터 조절 - svm 튜닝
svm_tune <- tune(svm, train.x = x, train.y = y, kernel='radial',
ranges = list(cost=10^(-1:2)), gamma=2^(-2:2))
# 10-fold cross validation : 교차 검정
svm_tune
# - sampling method: 10-fold cross validation
#
# - best parameters:
# cost
# 1
#
# - best performance: 0.03333333
svm_model_after_tune <- svm(Species ~ ., data=iris, kernel='radial', cost=1, gamma=0.5)
summary(svm_model_after_tune)pred <- predict(svm_model_after_tune, x)
t <- table(pred, y)
t
# y
# pred setosa versicolor virginica
# setosa 50 0 0
# versicolor 0 48 2
# virginica 0 2 48
sum(diag(t)) / nrow(x) # 0.9733333# KNN : 최근접 이웃 알고리즘을 사용. k개의 원 안에 데이터 수
# 또는 거리(유클디안 거리 계산법)에 의해 새로운 데이터를 분류.
# 비모수적 검정 분류 모형
install.packages("ggvis")
library(ggvis)
head(iris, 2)
iris %>% ggvis(~Petal.Length, ~Petal.Width, fill=~factor(Species))