클러스터링(Clustering)
: 비지도 학습의 일종
: 계층적 군집분석
- 계층적 군집분석 종류
- 응집형 : 자료 하나하나를 군집으로 간주하고, 가까운 군집끼리 연결하는 방법. 군집의 크기를 점점 늘려가는 알고리즘. 상향식
- 분리형 : 전체 자료를 큰 군집으로 간주하고, 유의미한 부분을 분리해 나가는 방법. 군집의 크기를 점점 줄여가는 알고리즘. 하향식
k-means : 군집 수(k) 지정. 거리(유클리디안 거리 계산 법)들의 평균으로 비계층적 군집분석 진행.
- 이론
군집 분석 (Clustering analysis)
군집 분석은 각 개체의 유사성을 측정하여 높은 대상 집단을 분류하고, 군집에 속한 개체들의 유사성과 서...
blog.naver.com
* cluster1.py
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
plt.rc('font', family='malgun gothic')
np.random.seed(123)
var = ['x', 'y']
labels = ['점0','점1', '점2', '점3', '점4']
x = np.random.random_sample([5, 2]) * 10
df = pd.DataFrame(x, columns = var, index = labels)
print(df)
''' x y
점0 6.964692 2.861393
점1 2.268515 5.513148
점2 7.194690 4.231065
점3 9.807642 6.848297
점4 4.809319 3.921175
'''
plt.scatter(x[:, 0], x[:, 1], c='blue', marker='o')
plt.grid(True)
plt.show()
from scipy.spatial.distance import pdist, squareform
dist_vec = pdist(df, metric='euclidean') # 데이터간 거리를 유클리디안 거리계산을 사용하여 측정
print('distmatrix :', dist_vec)
# [5.3931329 1.38884785 4.89671004 2.40182631 5.09027885 7.6564396 2.99834352 3.69830057 2.40541571 5.79234641]
print(squareform(dist_vec)) # 데이터를 테이블 형태로 변경
'''
[[0. 5.3931329 1.38884785 4.89671004 2.40182631]
[5.3931329 0. 5.09027885 7.6564396 2.99834352]
[1.38884785 5.09027885 0. 3.69830057 2.40541571]
[4.89671004 7.6564396 3.69830057 0. 5.79234641]
[2.40182631 2.99834352 2.40541571 5.79234641 0. ]]
'''
row_dist = pd.DataFrame(squareform(dist_vec))
print(row_dist)
'''
0 1 2 3 4
0 0.000000 5.393133 1.388848 4.896710 2.401826
1 5.393133 0.000000 5.090279 7.656440 2.998344
2 1.388848 5.090279 0.000000 3.698301 2.405416
3 4.896710 7.656440 3.698301 0.000000 5.792346
4 2.401826 2.998344 2.405416 5.792346 0.000000
'''from scipy.spatial.distance import pdist
distance=pdist(df, metric='euclidean') : 데이터간 거리를 유클리디안 거리계산을 사용하여 측정
from scipy.spatial.distance import squareform
squareform(distance) : 데이터를 테이블 형태로 변경
from scipy.cluster.hierarchy import linkage # 응집형 계층적 군집분석
row_clusters = linkage(dist_vec, method='ward') # method : complete, single, average, ..
print(row_clusters)
'''
[[0. 2. 1.38884785 2. ]
[4. 5. 2.65710936 3. ]
[1. 6. 5.45400408 4. ]
[3. 7. 6.64710151 5. ]]
'''
df = pd.DataFrame(row_clusters, columns=['클러스터1', '클러스터2', '거리', '멤버 수'])
print(df)
'''
클러스터1 클러스터2 거리 멤버 수
0 0.0 2.0 1.388848 2.0
1 4.0 5.0 2.657109 3.0
2 1.0 6.0 5.454004 4.0
3 3.0 7.0 6.647102 5.0
'''from scipy.cluster.hierarchy import linkage
linkage(distance, method='ward') :응집형 계층적 군집분석
method : complete, single, average, ...
- linkage API
docs.scipy.org/doc/scipy/reference/generated/scipy.cluster.hierarchy.linkage.html
scipy.cluster.hierarchy.linkage — SciPy v1.6.1 Reference Guide
For method ‘single’, an optimized algorithm based on minimum spanning tree is implemented. It has time complexity \(O(n^2)\). For methods ‘complete’, ‘average’, ‘weighted’ and ‘ward’, an algorithm called nearest-neighbors chain is imple
docs.scipy.org
from scipy.cluster.hierarchy import dendrogram
dendrogram(row_clusters, labels=labels)
plt.tight_layout()
plt.ylabel('유클리드 거리')
plt.show()from scipy.cluster.hierarchy import dendrogram
dendrogram(linkage값, labels=) : dendrogram 생성

- 계층적 클러스터 분류 결과 시각화
from sklearn.cluster import AgglomerativeClustering
ac = AgglomerativeClustering(n_clusters = 3, affinity='euclidean', linkage='ward')
labels = ac.fit_predict(x)
print('결과 :', labels) # 결과 : [0 2 0 1 0]from sklearn.cluster import AgglomerativeClustering
AgglomerativeClustering(n_clueters = 3, affinty='euclidean', linkage='ward') : 병합 군집 알고리즘
a = labels.reshape(-1, 1)
print(a)
'''
[[0]
[2]
[0]
[1]
[0]]
'''
x1 = np.hstack([x, a])
print('x1 :', x1)
'''
x1 :
[[6.96469186 2.86139335 0. ]
[2.26851454 5.51314769 2. ]
[7.1946897 4.2310646 0. ]
[9.80764198 6.84829739 1. ]
[4.80931901 3.92117518 0. ]]
'''
x_0 = x1[x1[:, 2] == 0, :]
x_1 = x1[x1[:, 2] == 1, :]
x_2 = x1[x1[:, 2] == 2, :]
plt.scatter(x_0[:, 0], x_0[:, 1])
plt.scatter(x_1[:, 0], x_1[:, 1])
plt.scatter(x_2[:, 0], x_2[:, 1])
plt.legend(['cluster0', 'cluster1', 'cluster2'])
plt.show()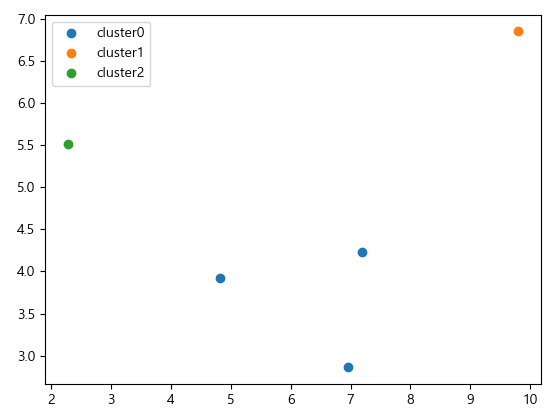
계층적 클러스터링 : iris
* cluster2.py
import pandas as pd
from sklearn.datasets import load_iris
import matplotlib.pyplot as plt
iris = load_iris()
iris_df = pd.DataFrame(iris.data, columns=iris.feature_names)
print(iris_df.head(3))
'''
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm)
0 5.1 3.5 1.4 0.2
1 4.9 3.0 1.4 0.2
2 4.7 3.2 1.3 0.2
'''
print(iris_df.loc[0:4, ['sepal length (cm)', 'sepal width (cm)']])
'''
sepal length (cm) sepal width (cm)
0 5.1 3.5
1 4.9 3.0
2 4.7 3.2
3 4.6 3.1
4 5.0 3.6
'''from scipy.spatial.distance import pdist, squareform
#dist_vec = pdist(iris_df.loc[:, ['sepal length (cm)', 'sepal width (cm)']], metric = 'euclidean')
dist_vec = pdist(iris_df.loc[0:4, ['sepal length (cm)', 'sepal width (cm)']], metric = 'euclidean')
print(dist_vec) # 데이터간 거리
# [0.53851648 0.5 0.64031242 0.14142136 0.28284271 0.31622777
# 0.60827625 0.14142136 0.5 0.64031242]row_dist = pd.DataFrame(squareform(dist_vec)) # 테이블 형태로 변경
print('row_dist :\n', row_dist)
'''
0 1 2 3 4
0 0.000000 0.538516 0.500000 0.640312 0.141421
1 0.538516 0.000000 0.282843 0.316228 0.608276
2 0.500000 0.282843 0.000000 0.141421 0.500000
3 0.640312 0.316228 0.141421 0.000000 0.640312
4 0.141421 0.608276 0.500000 0.640312 0.000000
'''from scipy.cluster.hierarchy import linkage, dendrogram
row_clusters = linkage(dist_vec, method='complete') # 응집형 계층적 군집 분석
print('row_clusters :\n', row_clusters)
'''
[[0. 4. 0.14142136 2. ]
[2. 3. 0.14142136 2. ]
[1. 6. 0.31622777 3. ]
[5. 7. 0.64031242 5. ]]
'''
df = pd.DataFrame(row_clusters, columns=['id1', 'id2', 'dist', 'count'])
print(df)
'''
id1 id2 dist count
0 0.0 4.0 0.141421 2.0
1 2.0 3.0 0.141421 2.0
2 1.0 6.0 0.316228 3.0
3 5.0 7.0 0.640312 5.0
'''
row_dend = dendrogram(row_clusters) # dendrodgram
plt.ylabel('dist test')
plt.show()
from sklearn.cluster import AgglomerativeClustering
ac = AgglomerativeClustering(n_clusters = 2, affinity='euclidean', linkage='complete')
x = iris_df.loc[0:4, ['sepal length (cm)', 'sepal width (cm)']]
labels = ac.fit_predict(x)
print('클러스터 결과 :', labels) # 결과 : [1 0 0 0 1]plt.hist(labels)
plt.grid(True)
plt.show()
비계층적 군집분석
yganalyst.github.io/ml/ML_clustering/
[클러스터링] 비계층적(K-means, DBSCAN) 군집분석
비계층적 군집분석 방법인 K-means와 DBSCAN에 대해 알아보자
yganalyst.github.io
'BACK END > Deep Learning' 카테고리의 다른 글
| [딥러닝] DBScan (0) | 2021.03.22 |
|---|---|
| [딥러닝] k-means (0) | 2021.03.22 |
| [딥러닝] Neural Network (0) | 2021.03.19 |
| [딥러닝] KNN (0) | 2021.03.18 |
| [딥러닝] RandomForest (0) | 2021.03.17 |




